Numpy (2)
N차원 배열의 선형 대수학
- Numpy에서 1차원, 2차원 및 n차원 배열의 차이점을 이해
- for 루프를 사용하지 않고 n차원 배열에 일부 선형 대수 연산을 적용하는 방법을 이해
- n차원 배열의 axis 및 shape 속성을 이해
# scipy.misc 모듈 의 face이미지를 사용
from scipy import misc
img = misc.face()
데이터 shape, axis, array 정보
img.ndim
3
# (세로, 가로, rgb)
img.shape
(768, 1024, 3)
# element의 수
img.size
2359296
img.dtype
dtype('uint8')
type(img)
numpy.ndarray
# 첫 픽셀의 rgb의 범위 출력
img[0, 0, :]
array([121, 112, 131], dtype=uint8)
print("값의 범위 : {} ~ {}".format(img.min(), img.max()))
값의 범위 : 0 ~ 255
import matplotlib.pyplot as plt
# %matplotlib inline 예전버전에서 이미지가 나오지 않을 경우 사용
plt.imshow(img)
plt.show() # imshow와 show 같은 cell에 사용

# r값 출력
img[:, :, 0]
array([[121, 138, 153, ..., 119, 131, 139],
[ 89, 110, 130, ..., 118, 134, 146],
[ 73, 94, 115, ..., 117, 133, 144],
...,
[ 87, 94, 107, ..., 120, 119, 119],
[ 85, 95, 112, ..., 121, 120, 120],
[ 85, 97, 111, ..., 120, 119, 118]], dtype=uint8)
# g값 출력
img[:, :, 1]
array([[112, 129, 144, ..., 126, 136, 144],
[ 82, 103, 122, ..., 125, 141, 153],
[ 66, 87, 108, ..., 126, 142, 153],
...,
[106, 110, 124, ..., 158, 157, 158],
[101, 111, 127, ..., 157, 156, 156],
[101, 113, 126, ..., 156, 155, 154]], dtype=uint8)
# img size 확인
img[:, :, 0].shape
(768, 1024)
- 0 - 255 이면 값의 차이가 크므로 0 - 1로 조정
차이가 크면 작은 값들의 중요도가 무시될 수 있기에
img_array = img / 255
print("값의 범위 : {} ~ {}".format(img_array.min(), img_array.max()))
값의 범위 : 0.0 ~ 1.0
# img 변화 확인
plt.imshow(img_array)
plt.show()

img_array.dtype
dtype('float64')
# 각 color channel을 별도의 array에 할당
red_array = img_array[:, :, 0]
green_array = img_array[:, :, 1]
blue_array = img_array[:, :, 2]
Axis에 작업
Numpy의 선형 대수 모듈인 numpy.linalg 를 사용하여 이 자습서의 작업을 수행한다. 이 모듈에 있는 대부분의 선형 대수 함수는 scipy.linalg 에서도 찾을 수 있으며 사용자는 실제 응용 프로그램에 scipy 모듈을 사용하는 것이 좋다. 그러나 SVD 함수와 같은 scipy.linalg 모듈의 일부 함수는 2D 배열만 지원한다.
-
- SVD (Singular Value Decomposition) 특이값 분해
- 특잇값 분해는 행렬을 특정한 구조로 분해하는 방식으로, 신호 처리와 통계학 등의 분야에서 자주 사용된다.
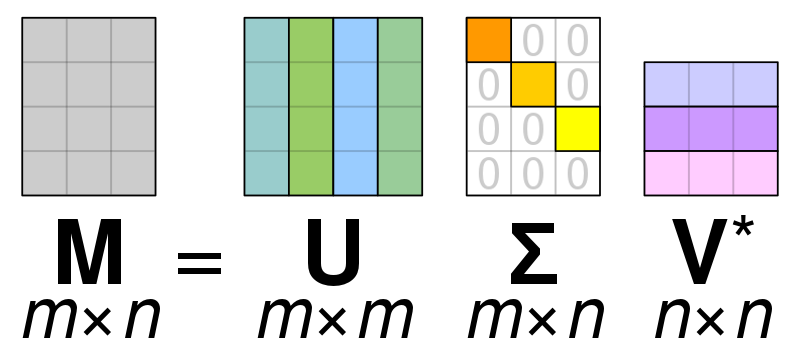
M (768x1024) =
U 전치행렬(768x768)
sigma 대각으로만 값 나머지 0(768,)
V^* 전치행렬(1024x1024)
모두 곱하면 원래값이 복원된다
# 선형대수 하위 모듈 불러오기
from numpy import linalg
rgb로 작업 시 복잡해지므로 grayscale로 바꿔서 작업(채널을 하나로 합침)
\[Y = 0.2126 R + 0.7152 G + 0.0722 B\]- array 곱셈 연산자
@를 사용
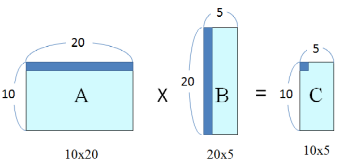
- 행렬곱의 전제조건: 앞쪽의 열과 뒤쪽의 행이 같아야 함
- 행렬곱의 결과: 앞쪽 array 행 x 뒤쪽 array 열
각 위치값들을 곱해서 더한다
img_gray = img_array @ [0.2126, 0.7152, 0.0722]
# (768, 1024, 3) @ (3, ) = (768, 1024)
img_gray.shape
(768, 1024)
plt.imshow(img_gray, cmap="gray") # 그냥 하면 임의 색으로 정해지므로 colormap지정
plt.show()

특이값 분해
# 특이값 분해
U, s, Vt = linalg.svd(img_gray)
U.shape, s.shape, Vt.shape
((768, 768), (768,), (1024, 1024))
# 행렬곱을 통해 원래대로 돌아오는지 확인
# 대각행렬의 shape를 조정해줘야 한다
import numpy as np
Sigma = np.zeros((U.shape[1], Vt.shape[0])) # fill_diagonal는 값을 채워주기만 하므로 초기화를 해 줘야 함 # (768, 1024)
np.fill_diagonal(Sigma, s) # 대각선으로 값을 채우기
Sigma[0:2]
array([[410.42098224, 0. , 0. , ..., 0. ,
0. , 0. ],
[ 0. , 85.56090199, 0. , ..., 0. ,
0. , 0. ]])
# 데이터 복원
(U @ Sigma @ Vt)[:2]
array([[0.45209882, 0.51876549, 0.57815529, ..., 0.47355843, 0.51387529,
0.54524784],
[0.33250118, 0.41485412, 0.49104706, ..., 0.46907059, 0.53181569,
0.57887451]])
근사치
linalg.norm(img_gray - U @ Sigma @ Vt) # 8가지 다른 매트릭스 노름 중 1가지를 반환 (벡터값들 사이의 차이값을 구하는 함수)
# 작을수록 근사치(거의 0에 근접: 원상복구 됨)
1.4108253216554015e-12
np.allclose(img_gray, U @ Sigma @ Vt) # 원래값과 차이가 없다면 True반환
True
plt.plot(s)
plt.show()
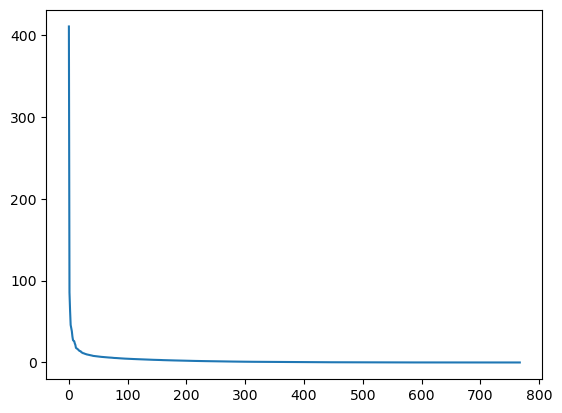
- s(sigma)의 대부분의 값이 매우 작음
k = 50 # 임의의 값
approx = U @ Sigma[:, :k] @ Vt[:k, :] # Sigma의 768개 행에 1024열중 10개만 사용, Vt의 1024열중 10개에 1024개 열 사용
plt.imshow(approx, cmap="gray")
plt.show()

- 분해해서 합칠 때 모든 정보가 필요한 것이 아니다! 일부만 있어도 가능
Color 이미지에 시도
r, g, b각각에 대해 위의 작업을 해도 되지만 numpy의 broadcast를 활용하면 쉽게 작업할 수 있다.
from PIL import Image
img_path = 'img01.jpg'
img = Image.open(img_path) # .convert('L') : grayscale로 불러옴
plt.imshow(img)
plt.show()
type(img)
PIL.JpegImagePlugin.JpegImageFile
# img를 ndarray로 변환
np.array(img)[:2]
array([[[120, 151, 180],
[120, 151, 180],
[120, 151, 180],
...,
[116, 149, 180],
[117, 150, 181],
[117, 150, 181]],
[[120, 151, 180],
[120, 151, 180],
[120, 151, 180],
...,
[114, 147, 178],
[115, 148, 179],
[116, 149, 180]]], dtype=uint8)
img2 = Image.open(img_path).convert('L')
plt.imshow(img2)
plt.show()

np.array(img2)[:2]
array([[145, 145, 145, ..., 143, 144, 144],
[145, 145, 145, ..., 141, 142, 143]], dtype=uint8)
# opencv 불러오기
import cv2
img3 = cv2.imread(img_path)
img3 = cv2.cvtColor(img3, cv2.COLOR_BGR2RGB) # opencv는 BGR로 불러오기에 RGB로 변환
img3[:2]
array([[[120, 151, 180],
[120, 151, 180],
[120, 151, 180],
...,
[116, 149, 180],
[117, 150, 181],
[117, 150, 181]],
[[120, 151, 180],
[120, 151, 180],
[120, 151, 180],
...,
[114, 147, 178],
[115, 148, 179],
[116, 149, 180]]], dtype=uint8)
plt.imshow(img3)
plt.show()
SVD를 적용할 array가 2차원 이상일 경우 모든 axis에 한번에 적용할 수 있다. 하지만 numpy의 선형 대수 함수는 (n, M, N) 형식의 array에 적용해야 한다. 이 형식의 첫번째 axis인 n은 M×N metrix가 얼마나 쌓여 있는지를 나타내는 숫자여야 한다.
img3.shape
(3200, 5220, 3)
img3_array = img3 / 255
img_array_transposed = np.transpose(img3_array, (2, 0, 1))
img_array_transposed.shape
(3, 3200, 5220)
# SVD 적용
U, s, Vt = linalg.svd(img_array_transposed)
U.shape, s.shape, Vt.shape
((3, 3200, 3200), (3, 3200), (3, 5220, 5220))
n차원 array 결과물
# s array가 곱셈이 가능한 형태로 변환
Sigma = np.zeros((3, 3200, 5220))
for j in range(3):
np.fill_diagonal(Sigma[j, :, :], s[j, :])
Sigma.shape
(3, 3200, 5220)
# 데이터 복원
reconstructed = U @ Sigma @ Vt
reconstructed.shape
(3, 3200, 5220)
reconstructed.min(), reconstructed.max()
(-1.4564213143873975e-14, 1.000000000000282)
img3_array.min(), img3_array.max()
(0.0, 1.0)
부동 소수점 오류가 누적되면 원본 이미지 범위를 약간 벗어난 값이 생성될 수 있다
# 부동 소수점 오류 제거
reconstructed = np.clip(reconstructed, 0, 1)
plt.imshow(np.transpose(reconstructed, (1, 2, 0)))
plt.show()
# 근사치(approximation) 수행
k = 50
approx_img = U @ Sigma[..., :k] @ Vt[..., :k, :] # ...은 생략을 의미한다
approx_img.shape
(3, 3200, 5220)
approx_img = np.clip(approx_img, 0, 1)
plt.imshow(np.transpose(approx_img, (1, 2, 0)))
plt.show()

Reference
- 이 포스트는 SeSAC 인공지능 자연어처리, 컴퓨터비전 기술을 활용한 응용 SW 개발자 양성 과정 - 심선조 강사님의 강의를 정리한 내용입니다.
- NumPy.org : NumPy
- Wikipedia : 특잇값 분해
- Seo Young Ki : [Algorithm] 연속 행렬 곱셈 (동적계획)
- 정은규 주간야옹이 배경화면

댓글남기기